[파이썬] Null값(NAN)이 있는 데이터 코사인 유사도(Cosine Similarity) 구하기
in Development on Python
Cosine Similarity for matrix with NANs using Python
코사인 유사도(Cosine Similarity)에서 Null값(NAN) 처리 하는 방법
- 코사인 유사도 식
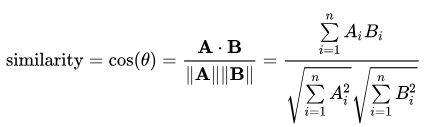
일단 코사인 유사도의 식은 간단한 것처럼 보인다. 그러나 이 아래 문제를 보면 절대 쉬운 식은 아니라는 걸 알 수 있다.
- 문제점

만약 이 데이터가 있다고 해보자. 그러면 우리는 공백(NAN)값에 대해서 0점을 줬다고 평가할 수 있을까? 그렇지 않다고 생각하는 사람이 많을 것이다. 그러나 파이썬 sklearn의 Coine Similarity는 일단 공백(NAN)값이 있는 데이터는 계산을 해주지않으며, 구글링을해보면 df.fillna(0)으로 0값으로 치환한 다음 계산을 하라고 되어 있다.
그러나 이 값은 전혀 다른 결과를 내보여주고 있다. 예를 들어 사용자A와 사용자B의 코사인 유사도를 공백(NAN)값으로 계산하게 된다면 값은 1이 나온다. 그러나 공백(NAN)을 0으로 치환해서 계산하게 되면 0.172라는 계산이 나온다. 다시말해 값에 모순이 생기게 된다.
그렇다면 파이썬에서 어떻게 공백(NAN)값을 포함해서 코사인 유사도(Cosine Similarity)를 계산할 수 있을 지 연구해보자.
1. NAN을 0으로 치환 후 코사인 유사도를 계산하는 방법
# 먼저 NAN이 포함되어 있는 3X4의 매트릭스를 만들어 보자
import numpy as np
nan_matrix = np.array([[3, np.nan, 2, 4],[1,2,3,4],[5,4,3,2]])
nan_matrix
array([[ 3., nan, 2., 4.],
[ 1., 2., 3., 4.],
[ 5., 4., 3., 2.]])
# DataFrame으로 만든다.
import pandas as pd
dataset = pd.DataFrame(nan_matrix, columns=['A','B','C','D'])
dataset
| A | B | C | D | |
|---|---|---|---|---|
| 0 | 3.0 | NaN | 2.0 | 4.0 |
| 1 | 1.0 | 2.0 | 3.0 | 4.0 |
| 2 | 5.0 | 4.0 | 3.0 | 2.0 |
# fillna(0)로 NAN값을 0으로 치환한다
nan_matrix_zero = dataset.fillna(0)
nan_matrix_zero
| A | B | C | D | |
|---|---|---|---|---|
| 0 | 3.0 | 0.0 | 2.0 | 4.0 |
| 1 | 1.0 | 2.0 | 3.0 | 4.0 |
| 2 | 5.0 | 4.0 | 3.0 | 2.0 |
# sklearn의 comsine similarity를 구할 수 있다.
from sklearn.metrics.pairwise import cosine_similarity
cos_sim_array = cosine_similarity(nan_matrix_zero)
cos_sim_array
array([[1. , 0.84757938, 0.73282811],
[0.84757938, 1. , 0.74535599],
[0.73282811, 0.74535599, 1. ]])
2. NAN이 있는 데이터 코사인 유사도를 계산하는 방법
# 같은 데이터로 치환 없이 코사인 유사도를 계산
nan_matrix
array([[ 3., nan, 2., 4.],
[ 1., 2., 3., 4.],
[ 5., 4., 3., 2.]])
def new_cosine(u, v):
m = u.shape[0]
udotv = 0
u_norm = 0
v_norm = 0
for i in range(m):
if (np.isnan(u[i])) or (np.isnan(v[i])): # Logical OR operator 참고
continue
udotv += u[i] * v[i]
u_norm += u[i] * u[i]
v_norm += v[i] * v[i]
u_norm = np.sqrt(u_norm)
v_norm = np.sqrt(v_norm)
if (u_norm == 0) or (v_norm == 0):
ratio = 1.0
else:
ratio = udotv / (u_norm * v_norm)
return ratio
# 몇개의 행으로 되있는지 그리고 3개를 array 형태 0,1,2으로 나눈다
len_array=np.arange(len(nan_matrix))
len_array
array([0, 1, 2])
# meshgrid를 이용해서 2차원 영역에 대한 좌표값을 만들어 준다. xx, yy를 만들어 준다.
xx, yy = np.meshgrid(len_array, len_array)
xx, yy
(array([[0, 1, 2],
[0, 1, 2],
[0, 1, 2]]), array([[0, 0, 0],
[1, 1, 1],
[2, 2, 2]]))
# dlwp zip을 이용해서 김밥을 자르듯 좌표값을 만들어 준다.
[list(zip(x, y)) for x, y in zip(xx, yy)]
[[(0, 0), (1, 0), (2, 0)], [(0, 1), (1, 1), (2, 1)], [(0, 2), (1, 2), (2, 2)]]
# 좌표값을 만들어서 new_cosine def를 쓰면 된다.
pd.DataFrame([new_cosine(np.array(nan_matrix[i]), np.array(nan_matrix[j])) for i, j in zip(x,y)] for y, x in zip(xx, yy))
| 0 | 1 | 2 | |
|---|---|---|---|
| 0 | 1.000000 | 0.910446 | 0.873589 |
| 1 | 0.910446 | 1.000000 | 0.745356 |
| 2 | 0.873589 | 0.745356 | 1.000000 |
